Datasets
El conjunto de datos consta de imágenes tomadas a través de un microscopio de células obtenidas tras realizar una biopsia de seno, para distintos pacientes (dentro de cada paciente las imágenes se encuentran a su vez agrupadas según subregiones). Está compuesto por aproximadamente 90000 pares de imágenes de histopatología de cáncer de mama teñidas con H&E y sus respectivas imágenes H-DAB, donde se tienen imágenes del estado frente a receptores que son usados como marcadores tumorales: receptor de estrógeno (ER), receptor de progesterona (PR), antígeno Ki-67, y oncogén HER2/neu.
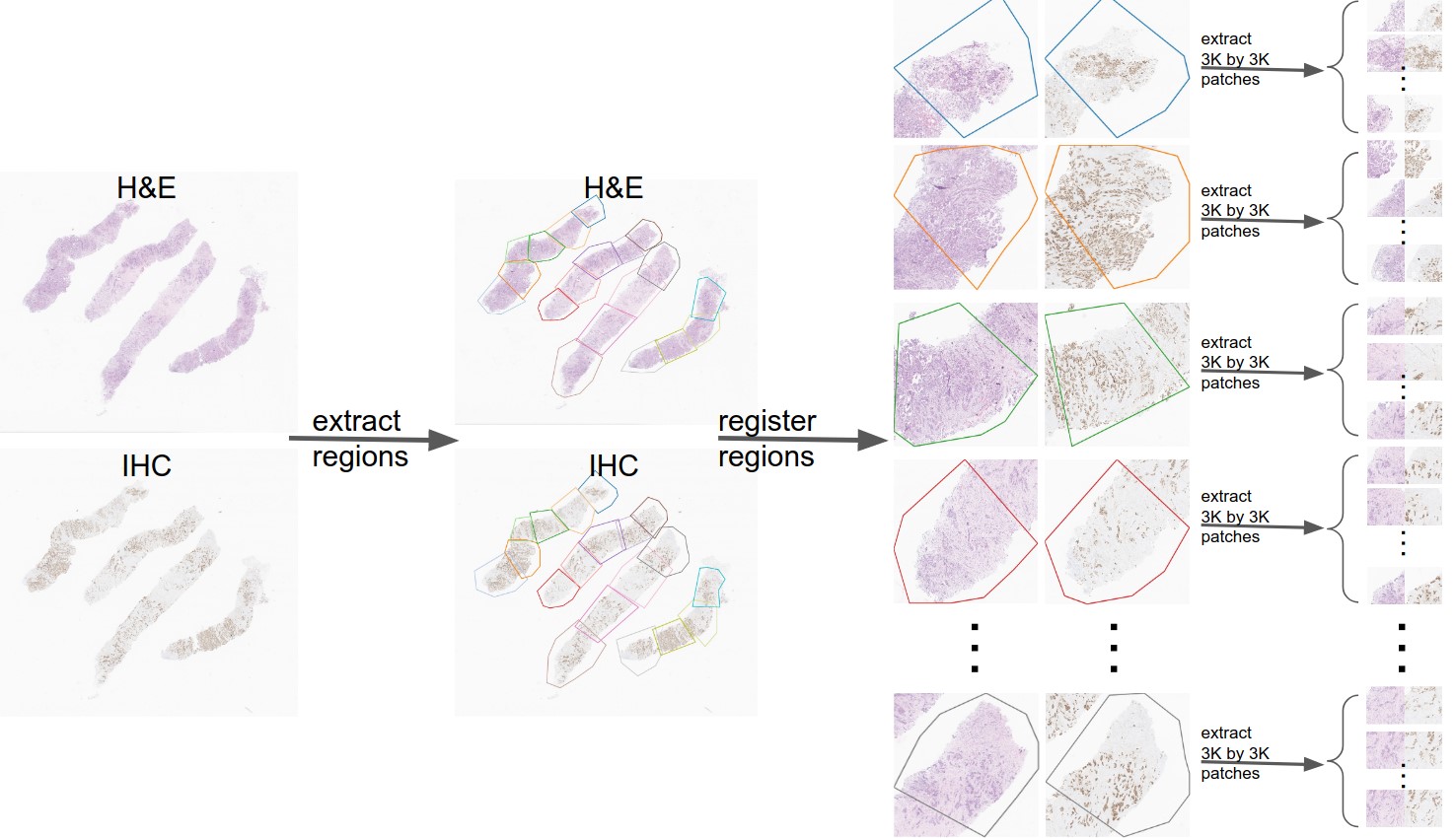
En la figura 1 se muestra un esquema de cómo se obtienen las distintas imágenes que conforman el conjunto de datos.
La tinción de hematoxilina y eosina (H&E) es una de las principales tinciones de tejido utilizadas en histología. La hematoxilina tiñe los núcleos celulares de un azul violáceo, y la eosina tiñe la matriz extracelular y el citoplasma de rosa, y otras estructuras adquieren diferentes tonos, matices y combinaciones de estos colores.
La DAB (diaminobencidina) se oxida en presencia de peroxidasa y peróxido de hidrógeno, lo que da lugar a la deposición de un precipitado marrón insoluble en alcohol en el sitio de la actividad enzimática. La DAB (diaminobencidina) produce un producto de reacción marrón oscuro y se puede utilizar tanto para aplicaciones inmunohistoquímicas como de transferencia. La DAB es eficaz como etiqueta única o como segundo color para el etiquetado de múltiples antígenos (como lo es en nuestro caso el Ki-67).
Para cada imagen H-DAB, el resultado del análisis DAB se proporciona en un archivo .csv correspondiente que incluye: el canal DAB promedio dentro de todos y cada uno de los núcleos en la imagen H-DAB, así como el número de núcleos en la imagen H&E. A partir del archivo .csv, se pueden calcular diferentes números como el puntaje H o el porcentaje de núcleos positivos. Esto último es lo que se realizó en nuestro caso, donde se implementó una función que calcula a partir de los datos el porcentaje de núcleos teñidos de marrón en las imágenes de reacción frente al Ki-67 (núcleos que están realizando proliferación celular y son por ende posibles células tumorales).
Para cada tipo de imagen H-DAB el archivo .csv correspondiente a cada paciente posee:
- file_image: nombre del archivo de la imagen a la que corresponden las labels
- manual_annot: anotación manual realizada por un experto que revisó todos los pares y asignó una de las etiquetas (a: el resultado del análisis DAB es confiable y se acepta, 0: la región es negativa para ese biomarcador, d: el par se descarta del conjunto de datos)
- num_nuclei_HandEsort: número de núcleos total presentes en la imagen
- avgDABnuclei_IHC: columna de largo variable con el promedio asignado a cada núcleo de nivel de marrón que tiene en la imagen. En los experimentos del artículo, los núcleos se estratifican en 0, 1+, 2+ y 3+ colocando determinados umbrales en el canal DAB promedio de cada núcleo (Ki-67: 0,12, 0,35, 0,55; ER: 0,06, 0,26, 0,46; PR: 0,06, 0,26, 0,46).
Dentro del conjunto de imágenes se tiene una gran variabilidad, existiendo aquellas que son consideradas médicamente ''correctas'' (la tinción se realizó bien, se pudo captar correctamente la respuesta al marcador), y otras que no aportan información, ya sea porque ocurrió una sobreexposición de la imagen (imágenes muy blancas), porque aparecen manchas por un error en la tinción, o porque la parte de composición de esa subregión o imagen en particular no posee una cantidad total de núcleos que aporte al análisis. Por ejemplo, en la figura 2 se tiene a la izquierda una imagen que es médicamente aceptable y a la derecha una que no lo es. En el proceso de análisis que implementamos se toma en cuenta tanto teóricamente como en código la existencia de ambos tipos de imágenes que aportan más o menos información.


Dataset utilizado
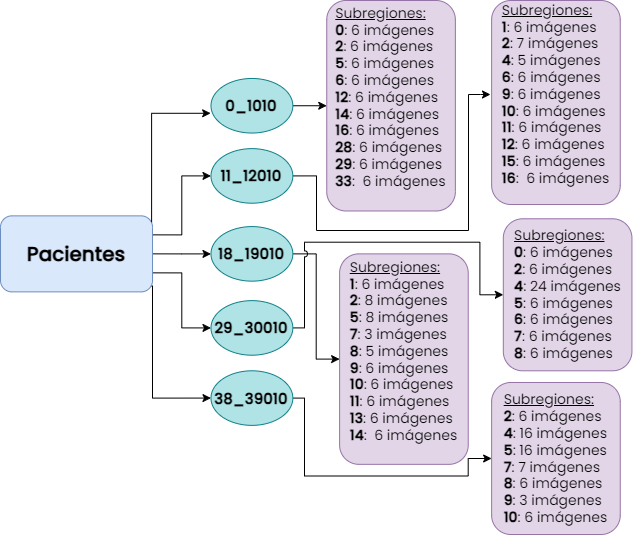
Dado el gran tamaño del dataset, trabajamos con una versión reducida del mismo creada por nosotros. Trabajamos con 5 pacientes, y para cada uno consideramos aproximadamente 10 subregiones, y luego aproximadamente 6 imágenes por cada subregión.
En algunos casos, no fue posible conseguir cumplir estos requerimientos, por lo que se compensó utilizando menos subregiones, pero más imágenes por subregión.
Se consideraron entonces 60 imágenes por cada uno de los cinco pacientes. Los mismos son identificados de la siguiente manera:
- Paciente 0_1010
- Paciente 11_12010
- Paciente 18_19010
- Paciente 29_30010
- Paciente 38_39010
Un esquema que muestra la composición del conjunto de imágenes utilizado se puede observar a continuación:
Este dataset reducido se puede encontrar en el siguiente link.